Einem internationalen Team von Wissenschaftlern ist es gelungen, einen virtuellen Röntgenlaser zu entwickeln, mit dessen Hilfe der „Heilige Gral“ der Strukturbiologie methodisch erschlossen werden kann.
Man stelle sich vor, man könnte mit einem Röntgenlaser ein individuelles Biomolekül auf atomarer Ebene aufnehmen, ohne dafür Kristalle zu züchten – ein Verfahren, das sehr zeitaufwendig und in manchen Fällen gar nicht möglich ist. Diese Idee des „single-particle imaging“ gilt als „Heiliger Gral“ der Strukturbiologie, der neue Möglichkeiten in vielen Forschungsfelder eröffnen würde, etwa im Bereich der Medikamenten-Entwicklung oder der ökologischen Forschung. Einem internationalen Team unter der Führung von Wissenschaftlerinnen und Wissenschaftlern des CFEL und des European XFEL ist es gelungen, eine erste detaillierte Umgebung für Computer-Simulationen für ein vollständiges Experiment an einem Freie-Elektronen Laser zu entwickeln. Eine Beschreibung der Software und ihrer Anwendungsmöglichkeiten wurde in der aktuellen Ausgabe der Scientific Reports veröffentlicht.
Citation:
C. H. Yoon, M. V. Yurkov, E..A. Schneidmiller, L. Samoylova, A. Buzmakov, Z. Jurek, B. Ziaja, R. Santra, N. D. Loh, T. Tschentscher, A. P. Mancuso
“A comprehensive simulation framework for imaging single particles and biomolecules at the European X-ray Free-Electron Laser”
Sci. Rep. 6 (2016) 24791
DOI: 10.1038/srep24791
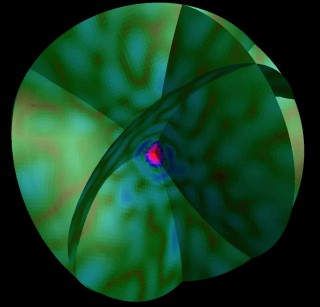
Illustration eines mit der simS2E Software erstellten zweidimensionalen Beugungsbildes, das mit Hilfe der Analysesoftware zu einem dreidimensionalen Bild zusammengefügt werden konnte. Foto: N. Duane Loh, National University of Singapore

