Mit einem leistungsstarken Röntgenlaser hat ein internationales Forscherteam erstmals einen Gen-Schalter in Aktion beobachtet. Die Untersuchung unter Leitung von Dr. Yun-Xing Wang vom US-Krebsforschungsinstitut (National Cancer Institute) zeigt die ultraschnelle Dynamik eines sogenannten Riboswitches, der einzelne Gene an- und ausschalten kann. Die angewendete Methode eröffnet neue Möglichkeiten zur Untersuchung zahlreicher grundlegender biochemischer Reaktionen, wie das Forscherteam im britischen Fachblatt „Nature“ berichtet. „Die Untersuchung belegt, dass sich die Strukturveränderungen, die bei biochemischen Reaktionen oder bei Wechselwirkungen zwischen Molekülen ablaufen, mit Hilfe leistungsfähiger Röntgenlaser in Echtzeit aufzeichnen lassen”, erläutert CUI-Prof. Henry Chapman (Universität Hamburg, DESY, CFEL), Ko-Autor der Studie.
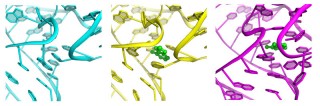
Der Riboswitch-‚Knopf‘ vor, während und nach (v.l.n.r.) dem Andocken des Signalmoleküls (grün). Bild: Yun-Xing Wang und Jason Stagno, National Cancer Institute
Die Wissenschaftler haben einen Riboswitch des Bakteriums Vibrio vulnificus untersucht. Dieses ist ein enger Verwandter des Cholera-Erregers und kann schwer zu behandelnde Infektionen auslösen, die häufig tödlich verlaufen. Der Riboswitch wird von einem Signalmolekül aktiviert, einem sogenannten Liganden – in diesem Fall ist das die Nukleinbase Adenin. Durch die Aktivierung ändert der Riboswitch seine Form. Das Gen, an dem er sich befindet, kann daraufhin nicht mehr abgelesen werden, ist also deaktiviert. Riboswitche gibt es vor allem bei Bakterien und Pilzen, nicht aber bei Säugetieren oder Menschen. Diese Gen-Schalter könnten daher aussichtsreiche Angriffspunkte im Kampf gegen Infektionskrankheiten sein.
Um zu beobachten, wie der Schalter aktiviert wird, kristallisierten die Forscher seinen „Einschaltknopf“, also den Part, an den das Signalmolekül Adenin bindet. Dieser Knopf ist wissenschaftlich gesprochen ein Aptamer. Das bedeutet, dass seine molekulare Struktur aus einer Sequenz von Nukleinsäuren besteht und nicht wie Proteine aus Aminosäuren. Die winzigen Aptamer-Kristalle schossen die Forscher in den extrem hellen Strahl des Röntgenlasers LCLS am US-Forschungszentrum SLAC in Kalifornien. Kristalle streuen Röntgenstrahlung auf charakteristische Weise, und aus dem Röntgenstreumuster lässt sich die Struktur des Kristalls und damit die seiner Bestandteile atomgenau berechnen. Daraus ergibt sich in diesem Fall die genaue räumliche Struktur des Aptamers.
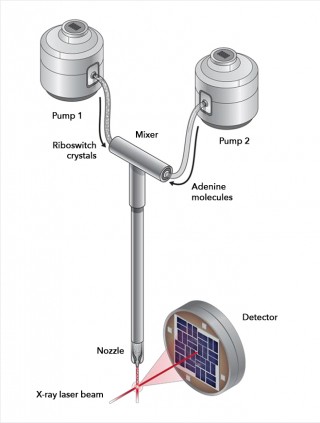
Neu entwickelter Versuchsaufbau: Das Röntgenstreubild (unten) liefert die atomare Struktur der Kristalle. Illustration: Joseph Meyer, National Cancer Institute
In einer neuen, von Chapmans Gruppe entwickelten Apparatur lassen sich die Aptamer-Kristalle mit einer Lösung der Adenin-Signalmoleküle mischen. Das Adenin verteilt sich dabei in den kleinen Kristallen und drückt so die Knöpfe der Riboswitche im Kristall. Die Adenin-getränkten Nanokristalle wurden in den Strahl des Röntgenlasers injiziert und analysiert. Eine Verzögerungsleitung ermöglichte es dabei, die biochemische Reaktion zu einer kurzen Zeit nach ihrem Beginn festzuhalten. Auf diese Weise entdeckten die Wissenschaftler einen Zwischenzustand des Gen-Schalters, der zuvor noch nie beobachtet worden war und der im lebenden Organismus vermutlich nur Millisekunden existiert. „Bisherige Experimente am SLAC-Röntgenlaser haben bereits biologische Reaktionen untersucht, die von Licht ausgelöst werden, wie etwa die Photosynthese. Dieses ist das erste Experiment, das in Echtzeit und auf der atomaren Skala eine Reaktion beobachtet hat, die von der chemischen Wechselwirkung zweier Biomoleküle ausgelöst wird“, erläutert Wang. „Dies illustriert die einzigartigen Möglichkeiten von Freie-Elektronen-Röntgenlasern, die so keine andere existierende oder absehbare Technologie bieten kann. Sie sind wie eine Kamera mit sehr kurzer Belichtungszeit, so dass sich jede Bewegung eines Biomoleküls in der Aktion einfangen lässt.“
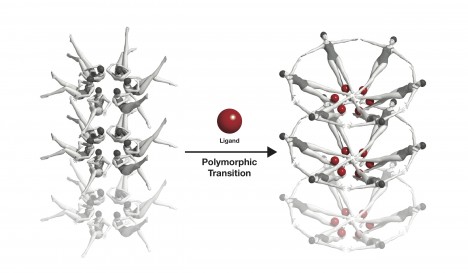
Trifft ein Ligand (rot) auf einen Riboswitch, ändert dieser seine Form. Diese Konformationsänderung ist hier symbolhaft mit Schwimmerinnen dargestellt. Illustration: Joseph Meyer, National Cancer Institute
Die Untersuchung zeigt, dass das Konzept der „Mix-and-Inject“-Kristallographie funktioniert, wie Chapman betont. Mit der Konformationsänderung der Aptamere änderte sich die Struktur und Symmetrie des gesamten Kristalls. Das geht nur bei sehr kleinen Kristallen, da größere durch die inneren Spannungen zerbrechen würden, die solche Formänderungen mit sich bringen. Außerdem kann sich nur in sehr kleinen Kristallen das Signalmolekül ausreichend schnell und gleichmäßig verteilen, so dass eine Untersuchung möglich wird. Derartige Nanokristalle erfordern extrem intensive Röntgenblitze zur Analyse, wie sie von Freie-Elektronen-Lasern wie LCLS oder dem European XFEL erzeugt werden, der zurzeit in der Metropolregion Hamburg in Betrieb genommen wird und dessen Hauptgesellschafter DESY ist.
„So gut wie alle Proteine, RNA und DNA interagieren mit Liganden oder Substraten und durchlaufen bestimmte Konformationsänderungen bei der Reaktion. Die Möglichkeit, diese Änderungen zu beobachten, ist eine Voraussetzung, um zu verstehen wie Biomakromoleküle ihre Funktion ausführen“, sagt Wang. „Bisher hat man diese Art von Reaktionen indirekt untersucht, bis auf sehr begrenzte Fälle, in denen die Konformationsänderungen sehr klein waren und den Kristall nicht zerbrochen haben. Mit unserer Methode können wir nun die Strukturen und Änderungen in einer viel breiteren Auswahl von biochemischen Wechselwirkungen und Reaktionen in Echtzeit beobachten.“
An der Studie beteiligt waren das US-Krebsforschungszentrum sowie weitere US-Gesundheitsinstitute, SLAC, DESY, die Arizona State University, die Johns-Hopkins-Universität, das Hauptmann-Woodward-Institut für medizinische Forschung, das US-Strukturbiologiezentrum und das US-Forschungszentrum Argonne National Laboratory.
Originalstudie:
J.R. Stagno, Y. Liu, Y.R. Bhandari, C.E. Conrad, S. Panja, M. Swain, L. Fan, G. Nelson, C. Li, D.R. Wendel, T.A. White, J.D. Coe, M.O. Wiedorn, J. Knoska, D. Oberthuer, R.A. Tuckey, P. Yu, M. Dyba, S.G. Tarasov, U. Weierstall, T.D. Grant, C.D. Schwieters, J. Zhang, A.R. Ferré-D’Amaré, P. Fromme, D.E. Draper, M. Liang, M.S. Hunter, S. Boutet, K. Tan, X. Zuo, X. Ji, A. Barty, N.A. Zatsepin, H. N. Chapman, J.C.H. Spence, S. A. Woodson & Y.-X. Wang
„Structures of riboswitch RNA reaction states by mix-and-inject XFEL serial crystallography“
Nature (2016)
DOI: 10.1038/nature20599
Weitere Informationen:
Zusamenfassung des National Cancer Institute
Mitteilung und Film des Forschungszentrums SLAC

