Ein Forscherteam von DESY und der Universität Hamburg erreicht einen weiteren Meilenstein auf dem Weg zur direkten Abbildung einzelner Biomoleküle: Die Gruppe unter der Leitung von Prof. Jochen Küpper vom Center for Free-Electron Laser Science hat eine neue Experimentiertechnik entwickelt, die es ermöglicht, verschiedene Strukturen eines Peptides voneinander zu trennen und sie so einzeln zu untersuchen und abzubilden. Über Ihre vielfältig einsetzbare Methode berichten die Forscher im Fachblatt „Angewandte Chemie“.
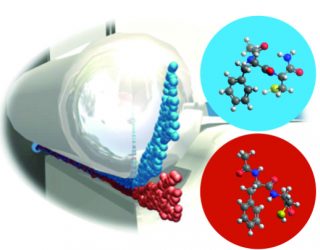
Unterschiedliche 3-D-Strukturen biologischer Moleküle, wie etwa ein Dipeptid mit zwei verschiedenen Konformeren (hier rot und blau abgebildet), können mit Hilfe eines kalten Molekularstrahls in einem starken elektrischen Feld getrennt werden. Die separierten Strukturen lassen sich dann einzeln untersuchen und abbilden. Illustration: UHH/N. Teschmit
Proteine gelten als Arbeitspferde des Lebens. Sie steuern die Funktion lebendiger Zellen und sind zum Beispiel für die Reproduktion von Zellen, aber auch für den Sauerstofftransport zuständig. Dieses weite Leistungsfeld verdanken sie ihrer einzigartigen, dreidimensionalen Struktur. Veränderungen in dieser Struktur können gravierende Auswirkungen auf die Funktion eines Proteins haben und sogar zu schwerwiegenden Erkrankungen führen. Die 3-D-Struktur eines Proteins wird dabei nicht nur durch die Sequenz der Aminosäuren beeinflusst, sondern auch von intramolekularen Wechselwirkungen, wie zum Beispiel Wasserstoffbrückenbindungen zwischen verschiedenen Teilen des Moleküls.
Ein gängiges Verfahren, um derartige Wechselwirkungen im Detail zu untersuchen, besteht darin, isolierte kleine Peptide (d.h. Ketten von einzelnen Aminosäuren) in der Gasphase zu untersuchen. Allerdings können selbst einzelne Aminosäuren und kleine Peptide unterschiedliche 3-D-Strukturen, sogenannte Konformere, einnehmen. Dieser Umstand erschwert die detaillierte Analyse dieser wichtigen biomolekularen Bausteine, da bildgebende Verfahren, wie zum Beispiel die Röntgenbeugung an Freie-Elektronen Lasern, Proben von identischen 3-D-Strukturen benötigen, um atomar aufgelöste Bilder zu erzeugen.
„Unser Ziel war es daher, neue Experimentiertechniken zu entwickeln, die Proben von Peptiden in der Gasphase erzeugen, die alle eine identische 3-D-Struktur aufweisen“, sagt Nicole Teschmit. Die Erstautorin der Studie forscht an der Universität Hamburg und ist wie ihre Kollegen Mitglied im Exzellenzcluster CUI. Mit der Laserdesorptions-Methode produzierte das Team zunächst sehr kalte (T ≈ 2 K) molekulare Strahlen intakter Dipeptid-Moleküle, welche sie anschließend mit Laserspektroskopie identifizierten. In diesem kalten Molekularstrahl können sich die verschiedenen Konformere nicht ineinander umwandeln. Um die verschiedenen Strukturen nun voneinander zu trennen, nutzen die Wissenschaftler starke elektrische Felder, welche mit den spezifischen Dipolmomenten der einzelnen Konformere wechselwirken und sie räumlich ablenken. Mit dieser Methode ist es den Forschern gelungen, die zwei Konformere des Dipeptids Ac-Phe-Cys-NH2 räumlich gänzlich voneinander zu trennen und reine Proben der einzelnen Konforme in der Gasphase zu erzeugen.
“Es ist uns erstmals gelungen, konformerselektierte Peptide in einem kalten Molekularstrahl zu demonstrieren. Diese Proben ermöglichen die Untersuchung von konformerspezifischen Prozessen mit allgemeinen Techniken, die sonst nicht zwischen verschiedenen Strukturen unterscheiden können“, sagt Co-Autor Dr. Daniel Horke. Die niedrige Temperatur des erzeugten Molekularstrahls ermöglicht es zudem, die Moleküle räumlich zu fixieren. Genau dies ist eine Voraussetzung für die Abbildung von Biomolekülen mit atomarer Auflösung, etwa am European XFEL. Küpper: „Unsere Methode ist ein Meilenstein auf dem Weg zur direkten strukturellen Abbildung individueller biologischer Moleküle.“ Text: CUI
Originalveröffentlichung:
Nicole Teschmit, Daniel A. Horke, Jochen Küpper
“Räumliche Trennung der Konformere eines Dipeptids”
Angewandte Chemie 130, 13971 (2018)
DOI: 10.1002/ange.201807646

